[本站讯]近日,控制科学与工程学院刘治平教授团队在蛋白质表面核酸绑定位点识别研究领域取得新进展,相关研究成果以“GeoBind: Segmentation of Nucleic Acid Binding Interface on Protein Surface with Geometric Deep Learning”为题,在线发表于Nucleic Acids Research (《核酸研究》,IF=19.16)。博士研究生李朋湃为第一作者,刘治平教授为通讯作者,山东大学为第一作者单位和通讯作者单位。
准确识别核酸绑定蛋白质(NBP)表面的结合位点有助于深入理解其在诸如基因转录调控、选择性剪接等细胞过程中的重要功能,利用蛋白质分子信息识别NBP绑定位点是结构生物信息学领域的重要问题,而蛋白质三级结构的有效表征是研究难点。针对上述问题,研究团队将蛋白质表面表示为点云(point clouds),开发利用几何深度学习模型以提取蛋白表面的核酸绑定位点的三维结构模式,提出了一种新型识别方法GeoBind,得到了准确的预测效果,比较研究表明该方法的识别精度显著超过当前SOTA(state-of-the-art)模型。
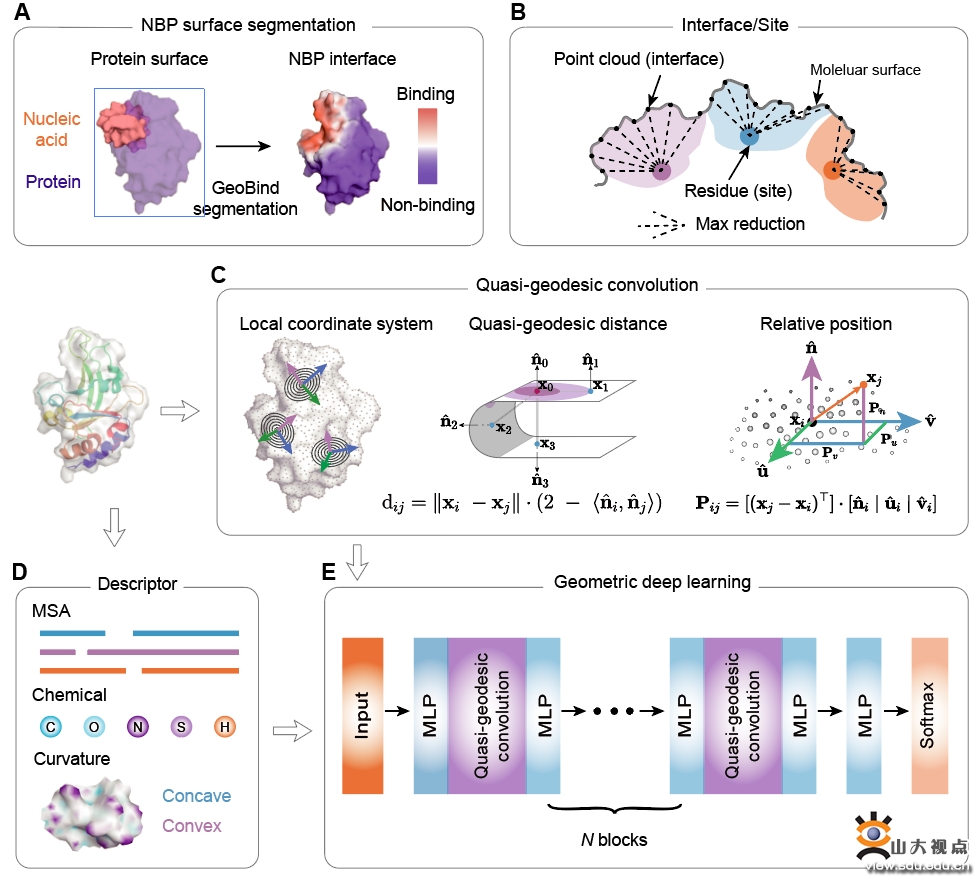
图 GeoBind方法流程示意图
该研究首次从蛋白质多聚体的角度,阐述了蛋白质从单体到多聚体构象变化对核酸绑定位点识别的影响,以及蛋白质从核酸未绑定状态到绑定状态下对绑定位点识别的影响。在近期人工智能大模型磅礴兴起的背景下,上述研究分析讨论了大规模预训练语言模型运用于蛋白质表征编码的可行性,研究成果为深入了解蛋白质与核酸相互作用机制提供了新的视角。
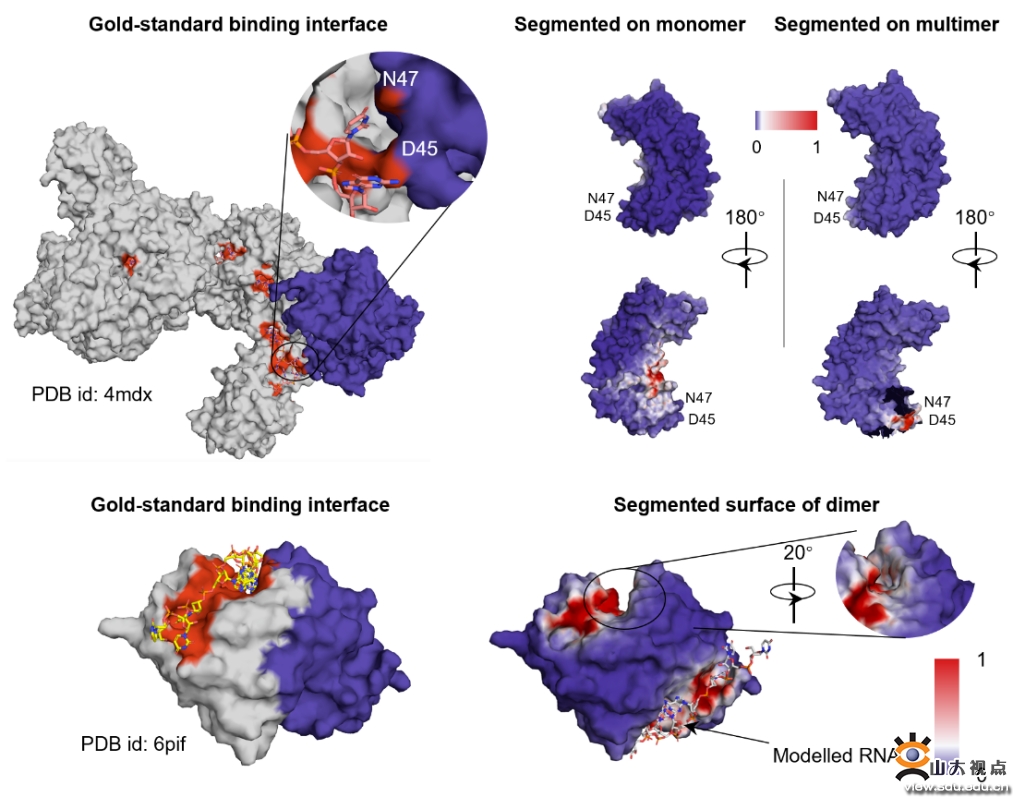
图 多聚体视角研究NBP绑定位点案例图
上述研究工作得到了国家重点研发计划、国家自然科学基金和山东大学集成攻关培育项目的大力支持。
原文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad288/7127225