[本站讯]近日,软件学院刘卫国教授团队在高性能分子动力学模拟软件研发方面取得进展,在高性能计算领域旗舰会议国际超算大会(SC)上发表了以“Cell-List based Molecular Dynamics on Many-Core Processors: A Case Study on Sunway TaihuLight Supercomputer”为题的研究论文。刘卫国教授的博士研究生段晓辉为该论文的第一作者,刘卫国教授为通讯作者。同时,该论文获得SC的最佳学生论文提名和最佳论文提名。这是我国第一次在SC获得最佳学生论文提名,也是我国第二次获得SC最佳论文提名。
图1.获得提名的论文信息
这项成果由刘卫国教授领导的软件学院高性能计算科研团队联合清华大学、国家超级计算无锡中心以及德国美因茨大学共同完成,刘卫国教授的博士生高萍、硕士生张萌和张庭坚也是该工作的主要贡献者。
分子动力学模拟是通过利用计算机求解体系内所有粒子的运动方程来模拟粒子的运动轨迹,从而获得系统的温度、体积、压力、应力等宏观和微观过程量。分子动力学模拟作为原子尺度模拟的经典计算方法,在计算物理、计算化学、材料科学、生命科学、生物医药等多个领域有着广泛的应用,已成为与实验同等重要的科学研究方法。分子动力学模拟过程中,需要花费大量时间用于计算范德华相互作用和静电相互作用,而模拟具有现实意义的生物大分子体系动力学过程往往需要几个月甚至几年时间。因此针对大规模体系开展大时间尺度的模拟,对范德华相互作用和静电相互作用的优化加速将是首要任务,其中进行软件和硬件加速是提高计算速度的重要方法。由美国D.E. Shaw Research(DESRES)设计的专门用于分子动力学模拟的超级计算机Anton2使用专用定制芯片进行加速,相较于通用型芯片,可将计算速度提高两个数量级。针对这一技术,美国对全球禁运,使得我国在相关领域的基础研究与应用开发速度和效率远远落后于美国。
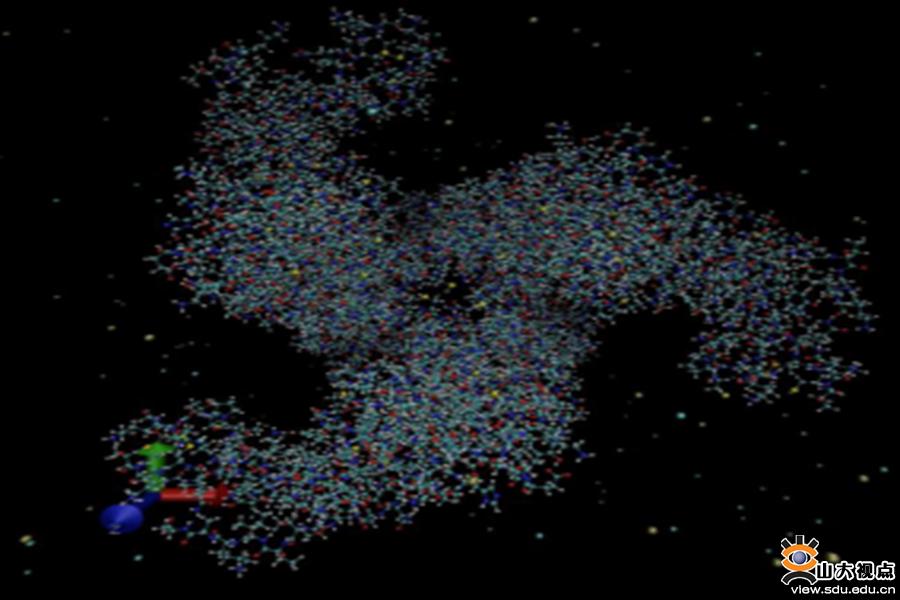
图2.使用ESMD模拟新冠病毒的木瓜蛋白酶样蛋白酶效果图
基于“神威·太湖之光”超级计算机的强大计算能力,项目团队成功地设计实现了高效、高可扩展的分子动力学模拟软件ESMD。ESMD以元胞列表取代传统的邻接表并通过对数据结构和算法的大幅重构在我国计算能力排名第一位的“神威·太湖之光”超级计算机上获得了显著的效率和扩展性的提升。实验表明ESMD在单个申威处理器上对AMBER的重新设计实现了50倍的性能提升并可在“神威·太湖之光”超级计算机的1024个节点上实现高达95%的弱扩展并行效率。这一基于国产处理器和超算平台的成果将极大地缩短我国与发达国家的在这一领域的差距,为深入正确地理解疾病的发生与发展、药物新靶标的发现与确认、疫苗研发、新材料和化学催化剂的研发提供有力的支撑,对稳定安全的支撑我国相关战略性产业的发展也具有重要意义。图2是使用ESMD对6W9C(新冠病毒木瓜蛋白酶样蛋白酶)模拟的效果图。本研究工作得到了科技部重点研发计划、国家自然科学基金和中德国际合作科研项目的支持。
论文链接:https://www.computer.org/csdl/proceedings-article/sc/2020/999800a295/1oeOS7D8uE8