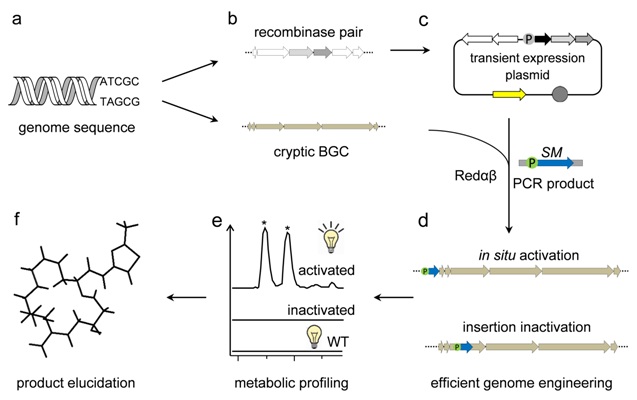
[本站讯]近日,山东大学微生物技术国家重点实验室团队在微生物基因组编辑和基因组发掘方面取得了新进展,建立了伯克氏菌中以Red/ET DNA重组工程技术为核心的基因组编辑和隐秘生物合成途径的原位激活平台。相关研究成果以“Discovery of recombinases enables genome mining of cryptic biosynthetic gene clusters in Burkholderiales species”(“发现新型重组酶介导的基因组编辑技术促进基因组发掘多种伯克氏菌中的隐秘生物合成基因簇”)为题,在线发表在国际著名期刊《美国科学院院报》(Proceedings of the National Academy of Sciences of the United States of America)上(Proc. Natl. Acad. Sci. U. S. A.,2018,115,DOI:10.1073/pnas.1720941115,五年影响因子达10.414)。该文章共同第一作者为微生物技术国家重点实验室博士研究生王雪、博士后周海波和联合培养博士生陈汉娜,山东大学微生物技术国家重点实验室为第一单位。
微生物是药物和生物农药的重要来源。全基因组测序发现微生物中仍然含有大量的隐秘生物合成基因簇尚未开发,隐秘基因簇编码的天然产物也是一个未被完全发掘的药物资源。利用启动子改造工程对激活这些基因簇,是一种有效的发掘隐秘基因簇的策略,而该策略的实施却受限于基因组编辑技术的匮乏,尤其是在基因簇丰富的非模式细菌中这种限制愈发明显。在本研究项目中,通过基因组扫描发现了一对新型的同源重组酶,并构建该重组酶的瞬时表达质粒将其应用到启动子改造工程激活未知基因簇中,因此在多株缺乏有效基因操作工具的非模式伯克氏菌中,建立了高效的基因组编辑技术;在此基础上实现了三株伯克氏菌的基因组发掘,激活和鉴定多个隐秘基因簇,并对两类新型的脂肽类化合物glidopeptins和rhizomides的结构和生物活性进行了系统研究。此外,本研究建立了一套有效伯克氏菌的基因组编辑技术,能够方便快捷地对多种伯克氏菌的基因组进行大片段删除和修饰工作,因此为构建天然产物异源表达的通用底盘菌提供了有力的技术支持。
本研究极大促进了伯克氏菌的基因组编辑和基因组发掘发现新型天然产物的发展,为深度利用和开发伯克氏菌资源奠定了技术基础。利用重组酶建立高效基因组编辑技术并激活基因簇的策略同样可以应用到其他细菌中,规模化地从细菌中发现具有良好活性的天然产物用于药物研发。而且本研究开辟了在天然产物产生菌中直接建立高效基因组编辑技术系统的研究,有利于在本源菌中激活沉默基因簇、直接编辑本源菌和生物组合合成等方法进行产量和结构优化等。
山东大学微生物技术国家重点实验室团队主要从事基于Red/ET同源重组工程的DNA大分子克隆和微生物基因组编辑技术体系的建立优化及产业化、以及以此为基础的微生物药物、转基因动物和病毒核酸疫苗研究领域的应用。团队核心成员卞小莹教授从事生物技术驱动的细菌基因组发掘和合成生物学研究,2015年入职以来,以第一作者/通讯作者发表论文7篇,主持省部级项目5项。
相关链接:http://www.pnas.org/content/early/2018/04/13/1720941115